近日,由经济学院统计学和数据科学系博士毕业生李晶茂与上海交通大学徐雅晴助理教授、耶鲁大学马双鸽教授、以及其导师方匡南教授合作完成的论文"GE-IA-NAM: Gene–environment Interaction Analysis via Imaging-assisted Neural Additive Model" 在Bioinformatics正式接受并在线发表,该论文是李晶茂博士论文的部分内容。李晶茂2024年7月毕业于厦门大学经济学院,现为耶鲁大学博士后。Bioinformatics为学界公认的统计学国际权威期刊,也是我院认定的统计学国际一类期刊(国际A-类)。
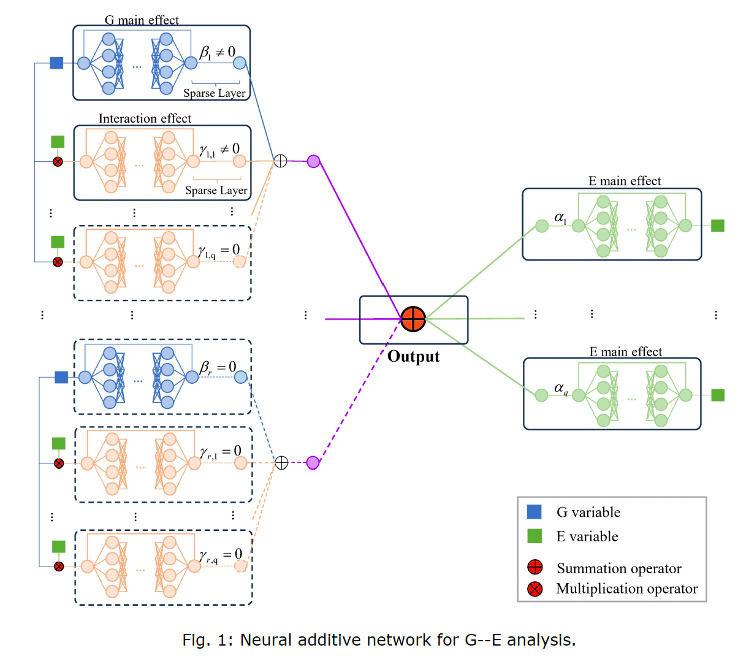
基因–环境(G–E)交互作用分析在健康医疗和公共卫生研究中具有重要意义,它有助于揭示基因变量与环境因素如何协同作用,从而影响疾病相关的结果变量。传统的G–E交互分析方法多依赖回归模型,但在处理复杂数据模式时往往显得力不从心。近年来,深度神经网络逐渐被引入这一领域,带来新的突破。然而,在样本量有限、数据维度高等“信息稀缺”情境下,这类方法的表现仍不尽如人意。值得注意的是,除了基因和环境数据,病理图像作为一种信息丰富且易于获取的数据来源,正逐渐展现出巨大的应用潜力。基于此,本文提出了一种多模态深度G–E交互学习框架,即基于病理图像辅助的神经网络可加模型(GE-IA-NAM),用于开展G–E交互作用分析。该方法通过加性网络结构,精细刻画基因变量、环境因素及其交互项对结果变量的影响。同时,研究团队设计了一种辅助学习策略,能够充分挖掘病理图像信息,有效提升建模效果。在模拟实验和实际数据分析中,该方法均展现出优异表现。与现有基于回归或全连接神经网络的G–E建模方法相比,新方法不仅在灵活性与可解释性之间取得更佳平衡,还能够在层次性约束下实现变量选择,显示出较强的创新性。此外,所提出的辅助学习策略也为多模态数据的统计分析提供了新的思路和方法论启示。